Il y a un an presque jour pour jour, Google Deepmind présentait Gnome, un modèle d’apprentissage par renforcement qui avait permis de découvrir plus de deux millions de structures cristallines stables, en surpassant les méthodes traditionnelles de simulation (lire Qant du 1er décembre 2023). Ces découvertes pourraient avoir des implications importantes, estimaient les chercheurs, dans la fabrication de batteries, la conception de matériaux pour l’électronique, ou encore dans l’élaboration de robots plus performants. Un an plus tard, c’est ce qui est en train de se passer.
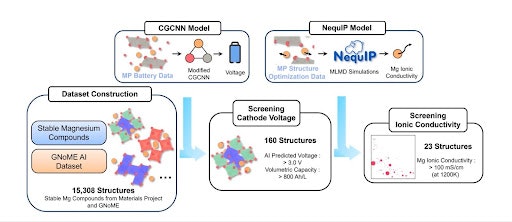
Un workflow entièrement basé sur l’IA aboutit à la découverte de 23 nouveaux matériaux pour des batteries • Source : Wenjie Chen et al.
Le petit chimiste, boosté à l’IA
Dans une étude pré-publiée la semaine dernière, des chercheurs de l'université Tsinghua ont développé un processus basé sur l'intelligence artificielle, dont Gnome, pour identifier de nouveaux matériaux de cathode pour batteries magnésium. En utilisant un réseau neuronal convolutif sur graphe (CGCNN), ils ont prédit avec précision les tensions de nombreux composés, sélectionnant 160 structures à haute tension. Un modèle NequIP (Neural Equivariant Interatomic Potentials) a ensuite permis de simuler la conductivité ionique, aboutissant à l'identification de 23 matériaux prometteurs pour des batteries magnésium à haute densité énergétique. Ce processus, qui accélère significativement la découverte de matériaux pour batteries, est entièrement basé sur l’IA.
En novembre, des chercheurs turinois ont présenté Energy-Gnome, une base de données de matériaux pour applications énergétiques, créée en exploitant la base de données GNoME pour identifier des matériaux prometteurs pour des applications thermoélectriques, des cellules photovoltaïques et des batteries. La base de données Energy-Gnome est conçue comme une ressource évolutive, alimentée par les contributions de la communauté scientifique.
De la chimie à la biologie
Paradoxalement, Demis Hassabis et John Jumper n’ont pas reçu le prix Nobel de chimie 2024 pour Gnome, mais pour Alphafold, un modèle conçu pour prédire la structure tridimensionnelle des protéines à partir de leurs séquences d'acides aminés. Le système a connu trois versions majeures depuis 2018. AlphaFold 1 a établi une première percée sur le repliement des protéines, tandis qu'AlphaFold 2 a introduit en 2020 une architecture entièrement nouvelle basée sur un modèle différenciable de bout en bout, atteignant une précision sans précédent dans la prédiction des structures protéiques.
La dernière version, AlphaFold 3, annoncée en mai dernier (lire Qant du 13 mai), étend les capacités du système au-delà des protéines individuelles. Elle peut désormais prédire les structures de complexes protéiques avec l'ADN, l'ARN, les modifications post-traductionnelles (les changements chimiques ou biologiques qui interviennent sur une protéine après sa synthèse initiale par le ribosome), ainsi que certains ligands et même des ions.
AlphaFold 3 utilise une nouvelle architecture appelée "Pairformer", inspirée de l’architecture Transformer des LLM, ainsi qu’un modèle de diffusion pour prédire les coordonnées atomiques, permettant une exploration plus diverse des solutions possibles. Un autre système d’IA de Google Deepmind, AlphaProteo, peut lui générer de nouvelles protéines.
Une multitude de Nobel et de modèles
Le troisième prix Nobel de chimie 2024, David Baker, est également un spécialiste de biologie “computationnelle”. Il est notamment connu pour les modèles d’IA Rosetta Fold et RF Diffusion. RosettaFold utilise des techniques d'apprentissage profond pour déterminer les structures des protéines à partir des séquences d'acides aminés. RosettaFold diffusion (RFdiffusion) incorpore des modèles probabilistes de diffusion par débruitage (DDPM) pour permettre la création de diverses protéines fonctionnelles à partir de spécifications moléculaires simples. Elle a été utilisée pour concevoir des centaines de nouvelles protéines aux fonctions diverses.
La conception générative de protéines est également la spécialité d’un autre grand modèle, ESM3, développé par EvolutionaryScale, une start-up fondée par d'anciens chercheurs de Meta AI. Ce modèle, qui compte 98 milliards de paramètres, a été entraîné sur 2,78 milliards de séquences de protéines provenant de divers organismes et biomes. Il est conçu pour raisonner simultanément sur la séquence, la structure et la fonction des protéines, utilisant une puissance de calcul d'un billion de téraflops. Il a notamment généré une protéine fluorescente verte (esmGFP), qui ne partage que 58% d'identité de séquence avec les protéines fluorescentes connues. Il a également été utilisé pour des enzymes d'intérêt, comme la PETase, qui dégrade le plastique PET. De plus, il peut simuler des processus évolutifs s'étendant sur des centaines de millions d'années.
Text-to-protein
Dès février 2023, des chercheurs américains ont présenté Protein-DT, un modèle qui permet de générer des protéines à partir d’une description textuelle. Les protéines deviennent une modalité, tout comme le texte, les images ou la vidéo. ESM3 va plus loin, en intégrant la séquence (la succession d'acides aminés), la structure (les coordonnées atomiques tridimensionnelles) et la fonction d’une protéine (son rôle biologique) dans un espace latent unifié.
Cette approche multimodale ouvre des perspectives pour l'ingénierie des protéines et la découverte de médicaments comparable à celle que l’on observe dans la chimie. Dans le nouveau domaine de la biologie programmable, les chercheurs peuvent désormais spécifier des détails fonctionnels de haut niveau, des exigences structurelles et des contraintes de séquence pour générer de nouvelles protéines.
Cette approche devrait accélérer la découverte de médicaments, en réduisant le temps et les coûts de développement de nouvelles thérapies. D’autant qu’ESM3 montre également une bonne compréhension des séquences et structures d'anticorps, permettant des opérations in silico comme la diversification et l'optimisation. D’autres modèles, non encore validés en laboratoire, préparent des approches spécialisées : Goab a été entraîné exclusivement sur des anticorps, Pepflow sur des peptides…
La route est longue de la découverte/conception de nouvelles molécules à la production de nouveaux médicaments, mais à l’horizon, la vague gonfle. Il serait surprenant qu’un raz-de-marée de nouvelles molécules ne s’abatte pas sur les laboratoires pharmaceutiques, issu aussi bien de la chimie que de la biologie. À partir de 2025, peut-on espérer.
Chimie + biologie = médecine
Or, dans le domaine médical, l’IA s’impose progressivement dans tous les domaines. Elle a tout d’abord été utilisée comme un outil pour améliorer le diagnostic. SynthSR, par exemple, un outil conçu pour la transformation des IRM, convertit des images de faible qualité en haute résolution. Cette innovation permet de standardiser les résultats des scanners cérébraux, améliorant ainsi l’identification de pathologies complexes comme les maladies neurodégénératives ou les tumeurs. Grâce à cet outil, les cliniciens peuvent mieux interpréter les données, même dans les régions où les appareils d’imagerie sont moins performants.
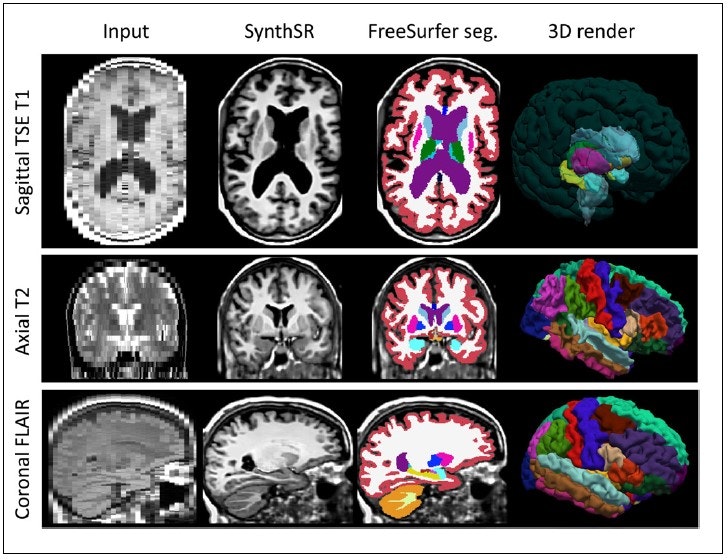
Générations de scanners cérébraux par SynthSR • Source: Iglesias et al., 2023
L’impact de l’IA s’étend également aux plateformes de traitement des données médicales. Les modèles comme GPT-4 Medprompt atteignent des niveaux de précision comparables à ceux des spécialistes humains, notamment dans les tests de diagnostic clinique. Ces systèmes deviennent des assistants précieux pour les médecins, surtout dans les zones sous-équipées où l’accès à des experts est limité.
En matière de santé publique, le modèle EVEscape anticipe les mutations des virus pour guider la recherche vaccinale. Cet outil a démontré sa capacité à prédire près de la moitié des mutations observées lors de l’évolution du SARS-CoV-2. En fournissant ces informations aux chercheurs, EVEscape contribue à accélérer les efforts pour concevoir des vaccins plus efficaces face aux futures menaces sanitaires.
Retour à Deepmind
Et pour ce qui est de la recherche, AlphaMissense, un modèle d'IA dérivé d’Alphafold, a prédit la pathogénicité de toutes les substitutions possibles d’un acide aminé unique dans le génome humain. En prédisant si chacune de ces 71 millions de variation est bénigne ou pathogène, le modèle a créé une ressource nouvelle pour la compréhension des maladies génétiques.
Ce foisonnement d’IA s’étend au-delà de la chimie, la biologie et la médecine. On le retrouve dans presque toutes les grandes disciplines scientifiques, signe que l’IA générative sature le champ sémantique humain. Il est souvent, quoique non exclusivement, basé sur les modèles de Deepmind, qui a également appliqué la même technologie d’apprentissage par renforcement à la météo, à la prédiction des inondations… et bien sûr à l’informatique.
AlphaDev, par exemple, a permis d’améliorer les algorithmes de tri et de réduire le nombre d’instructions nécessaires, augmentant ainsi l’efficacité des processus computationnels. C’est la première mise à jour significative des bibliothèques C++ en plus de dix ans, et cela pourrait avoir un impact direct dans des domaines comme la finance ou la cybersécurité, où la vitesse de calcul est essentielle.
AlphaDev est dérivé d’Alphazero, le seul modèle qui, jusqu’à présent, a prouvé qu’il était supérieur aux êtres humains, du moins quand il s’agit de jouer aux échecs ou au go.
Jusqu’à présent.
Pour en savoir plus :
- HAI Stanford University
- Wenjie Chen et al., AI-Driven Accelerated Discovery of Intercalation-type Cathode Materials for Magnesium Batteries, Arxiv 2024
- Paolo De Angelis et al., Energy-GNoME: A Living Database of Selected Materials for Energy Applications, Arxiv 2024
- Juan Iglesias et al., SynthSR: A public AI tool to turn heterogeneous clinical brain scans into high-resolution T1-weighted images for 3D morphometry, Science 2023
- La France et les robots, I : nos prévisions pour 2025
- La France et les robots, II : le défi mondial de gouverner l’IA
- La France sans les robotaxis : prévisions 2025, III
- À quand le grand chamboulement monétaire ? • prévisions 2025, IV
